Note: 這篇是去年二月寫在臉書專頁上的,不知道為什麼忘了移到這邊。總之,過了一年之後,有新的 update。
同卵雙生兄弟 Mark Kelly 和 Scott Kelly 兩位都是太空人,他們在兩年前參加了一個太空基因計畫(US$1.5-million),看看人類在太空無重力狀態時,DNA 和體內腸道菌會產生什麼變化。
Mark 在 2011 年退休前共在太空中待了 54 天,Scott 則是約 180 天,但是他在之後的 2015-16 年間(連續)待在太空中 340 天,合計一生中在太空待了 520 天。在 Scott 待在太空的那一年間,Mark 則待在地球,兩兄弟在 Scott 飛上太空之前、在太空中和回到地球後,都會固定收集血液、口水、尿液和糞便樣本,之後兩人全部的基因體(genome)都會被完整定序(但因為個人隱私所以只有十個科學家能取得定序結果)。
結果發現 Scott 在太空中的時候 telomere 變得比較長(比在地球的 Kelly 的還長),但是回到地球後,會迅速變短,回到他飛太空之前的長度。(telomere 是染色體兩端的蓋子,隨著年齡增長會越變越短。)科學家目前不知道為什麼會這樣,因為他們本來預期在太空中會變短,所以也正在研究另外十位太空人的 telomere,看是不是所有太空人都出現這種情形,預計 2018 年會知道結果。(哦哦~ 所以這是說在太空中會很長壽的意思嗎?XD)
另外就是 DNA methylation 在太空中的時候也漸少了,但是同時間在地球的 Mark 的 DNA methylation 卻增加了,而在 Scott 回到地球後,兩人的 DNA methylation 又回到 Scott 飛太空前的程度,為什麼會這樣還不知道。(這好神奇~)
"Personalized medicine could play into NASA’s plans for how to keep astronauts healthy during long-duration spaceflight, such as any future trips to Mars."
所以人類是已經準備好要進軍火星了嗎?XD
March 2018 update:
NASA 在今年一月發表了新的報告,他們發現 Scott 在太空時改變的基因表現中,雖然大多數(93%)在回到地球後又變回和飛太空之前一樣,但是有 7% (約幾百個)的基因表現並沒有變回去,這些基因和免疫系統(immune system)、DNA 修復(DNA repair)、骨頭形成(bone formation networks)、 缺氧(hypoxia)和高碳酸血症(hypercapnia)相關。
References
Alexandra Witze, Astronaut twins study raises questions about genetic privacy. Nature (2015)
Alexandra Witze, Astronaut twin study hints at stress of space travel. Nature (2017)
NASA / NASA Twins Study Investigators to Release Integrated Paper in 2018 (Jan 2018)
NASA / NASA Twins Study Confirms Preliminary Findings (Jan 2018)
2018年3月11日 星期日
2018年2月4日 星期日
關於抗體和抗體藥的一些小知識
近幾年來抗體藥變得頗為流行,不少藥廠研發抗體藥,其中包括治療阿茲罕默症的 aducanumab 和 solanezumab。藥名後面為 -mab 的代表是抗體藥,-mab 即為 mAb (monoclonal antibody)。

Figure / Wiki - Drug nomenclature(點圖可以放大)
學名藥命名方式可參考這:Generic Name Stems
大家都知道抗體 IgG 長什麼樣子吧?就是兩個 heavy chains 和兩個 light chains,辨認抗原(antigen)的那端是 Fv (variable regions),另一端則是會被醣化(glycosylation)和引發免疫反應的 Fc (constant regions)。但是,你知道嗎?駱駝(camelid, llama)的體內有兩種抗體哦哦哦!一種是跟人類一樣的 IgG,另一種是只有 heavy chains 的抗體(HCAbs),這種特別的抗體的 Fv 叫做 VHH (下圖),又稱 nanobodies (Nbs),雖然只有 heavy chain,但是對抗原的 binding affinity 並沒有比較弱,加上體積小,可以用來做抗體藥。

Figure / P. Holier & P. J. Hudson, Nature Biotechnology 2005 (doi: 10.1038/nbt1142)
簡單來說抗體藥有幾種,一種是整個 IgG,一種是只有 Fab,或甚至是只有 Fv,近來更小的 single-chain Fv (scFv) 和 VHH (Nbs) 也引起關注,這兩種小抗體有個好處,就是比完整的 IgG 更容易穿透細胞,也容易被清洗掉(這點有好也有壞,要看用在哪),而且因為小又沒有 Fc,所以打入體內做為治療時不會引起不必要的免疫反應。
目前做抗體藥的方法有分三種,一種是大家比較知道的 in vivo,先打到動物裡,再取出 B cells 和 myeloma 合成 hybridomas 生產 monoclonal antibody。另一種是在 E. coli 裡面製造,這種大家應該也知道,在細菌裡面製造的優勢就是量大且便宜。最後一種是在酵母菌裡面製造,在酵母菌製造的優勢也是量大且便宜,但比細菌好的是它是 eukaryotic。
因為正常情況下,Fc 的部分會醣化,所以如果要做整個 IgG 的話,最好是在 mammalian cells 裡面製造,就是用 hybridomas,因為細菌並沒有 glycosylation,所以如果用細菌的話,還要再另外 in vitro glycosylation,這麼麻煩的話為什麼還要用細菌呢?如果你是要製造 Fab 或是 scFv 的話,因為不需要 glycosylation,這時候用細菌就可以省時省錢。
酵母菌的話,則是可以用來製造 IgG 或是 Fab, scFv。但是酵母菌的 glycosylation 和人類的不一樣,酵母菌的只有 mannose,生產出來的會附帶大量的 mannose,但是人類的是比較複雜的,通常不只有 mannose,還有 galactose 和 sialic acids 等等,所以需要改造酵母的基因,讓它能夠生產 galactose 和 sialic acid。所以同樣的,如果只是製造 Fab 或是 scFv 的話,用酵母菌會比用動物便宜。如果要製造整個 IgG 的話,雖然比較複雜一點,但是如果基因調控好了,除了可以大量生產外,還可以控制抗體的品質,省錢省時間。另外就是利用酵母菌的話,生產的蛋白質可以讓它釋放出來到培養液中,這樣純化起來會比較容易。

Figure / Jens Nielsen, Bioengineered 2013 (doi: 10.4161/bioe.22856)
當然用 E. coli 或酵母菌的話,不只能製造抗體藥,其他的蛋白質藥物也可以,例如目前市面上已有用酵母菌生產的胰島素(by Novo Nordisk)。
有興趣的可以看下面幾篇 papers。
[05.12.2018 update]
這次 Science 發了這篇文章介紹我之前講的駱駝和鯊魚的小抗體。駱駝和鯊魚除了有正常的 IgG 抗體外,還有一種小抗體,是只有 heavy chain 的抗體。這種小抗體有跟正常抗體一樣的功能,同樣能抓住抗原(antigen),是免疫系統的一部份,在外來物體入侵時,體內會產生免疫反應製造出這些小抗體。小抗體的 variable region,也就是 VHH,又稱 nanobodies,近年被廣泛研究,看是否能用在治療疾病上面。Nanobody 的其中一種用法就是在它的一端接上讓蛋白降解訊息片段(signal sequence),例如 degron,這樣當 nanobody 抓到致病蛋白後,接著就會被帶去 proteasome 或是 lysosome 進行降解。
相關舊文:降解蛋白質的密碼(degron)
小抗體的發現是個意外,VUB 的學生想要分析血液和疾病的關聯的時候,因為不想殺老鼠取血,便想到了冷凍庫有駱駝寫的庫存,拿來用後卻發現駱駝血裡面除了有正常的 IgG,還有一種小型的抗體,研究後發現是沒有 light chain 的抗體。之後英國的博士生想要研究駱駝的小抗體,便請 VUB 的研究人員寄血液檢體砍他,沒想到在運送過程中血液莫名弄丟了。不過呢,之後他發現鯊魚血液裡也有這種只有 heavy chain 的小抗體。(這真是太神奇了,為什麼研究會從駱駝跳到鯊魚呢?)
Nanobody 也讓 Kobeilka 得到 2012 年的諾貝爾化學獎,Kobeilka 研究的是 β2-adrenergic receptor,他利用 nanobodies 把 receptor 鎖在 active state 以研究其 crystal structure,他把結果發表在 2011 年的 Nature,之後便在 2012 年因這項研究得到諾貝爾獎。
References:
Science / Mini-antibodies discovered in sharks and camels could lead to drugs for cancer and other diseases (May 2018)
S. Steeland et al, Nanobodies as therapeutics: big opportunities for small antibodies. Drug Discovery Today (2016)
P. Holliger & P.J. Hudson, Engineered antibody fragments and the rise of single domains. Nature Biotechnology (2005)
Rasmussen et al, Structure of a nanobody-stabilized active state of the β2 adrenoceptor. Nature (2011)

Figure / Wiki - Drug nomenclature(點圖可以放大)
學名藥命名方式可參考這:Generic Name Stems
大家都知道抗體 IgG 長什麼樣子吧?就是兩個 heavy chains 和兩個 light chains,辨認抗原(antigen)的那端是 Fv (variable regions),另一端則是會被醣化(glycosylation)和引發免疫反應的 Fc (constant regions)。但是,你知道嗎?駱駝(camelid, llama)的體內有兩種抗體哦哦哦!一種是跟人類一樣的 IgG,另一種是只有 heavy chains 的抗體(HCAbs),這種特別的抗體的 Fv 叫做 VHH (下圖),又稱 nanobodies (Nbs),雖然只有 heavy chain,但是對抗原的 binding affinity 並沒有比較弱,加上體積小,可以用來做抗體藥。

Figure / P. Holier & P. J. Hudson, Nature Biotechnology 2005 (doi: 10.1038/nbt1142)
簡單來說抗體藥有幾種,一種是整個 IgG,一種是只有 Fab,或甚至是只有 Fv,近來更小的 single-chain Fv (scFv) 和 VHH (Nbs) 也引起關注,這兩種小抗體有個好處,就是比完整的 IgG 更容易穿透細胞,也容易被清洗掉(這點有好也有壞,要看用在哪),而且因為小又沒有 Fc,所以打入體內做為治療時不會引起不必要的免疫反應。
目前做抗體藥的方法有分三種,一種是大家比較知道的 in vivo,先打到動物裡,再取出 B cells 和 myeloma 合成 hybridomas 生產 monoclonal antibody。另一種是在 E. coli 裡面製造,這種大家應該也知道,在細菌裡面製造的優勢就是量大且便宜。最後一種是在酵母菌裡面製造,在酵母菌製造的優勢也是量大且便宜,但比細菌好的是它是 eukaryotic。
因為正常情況下,Fc 的部分會醣化,所以如果要做整個 IgG 的話,最好是在 mammalian cells 裡面製造,就是用 hybridomas,因為細菌並沒有 glycosylation,所以如果用細菌的話,還要再另外 in vitro glycosylation,這麼麻煩的話為什麼還要用細菌呢?如果你是要製造 Fab 或是 scFv 的話,因為不需要 glycosylation,這時候用細菌就可以省時省錢。
酵母菌的話,則是可以用來製造 IgG 或是 Fab, scFv。但是酵母菌的 glycosylation 和人類的不一樣,酵母菌的只有 mannose,生產出來的會附帶大量的 mannose,但是人類的是比較複雜的,通常不只有 mannose,還有 galactose 和 sialic acids 等等,所以需要改造酵母的基因,讓它能夠生產 galactose 和 sialic acid。所以同樣的,如果只是製造 Fab 或是 scFv 的話,用酵母菌會比用動物便宜。如果要製造整個 IgG 的話,雖然比較複雜一點,但是如果基因調控好了,除了可以大量生產外,還可以控制抗體的品質,省錢省時間。另外就是利用酵母菌的話,生產的蛋白質可以讓它釋放出來到培養液中,這樣純化起來會比較容易。

Figure / Jens Nielsen, Bioengineered 2013 (doi: 10.4161/bioe.22856)
當然用 E. coli 或酵母菌的話,不只能製造抗體藥,其他的蛋白質藥物也可以,例如目前市面上已有用酵母菌生產的胰島素(by Novo Nordisk)。
有興趣的可以看下面幾篇 papers。
[05.12.2018 update]
這次 Science 發了這篇文章介紹我之前講的駱駝和鯊魚的小抗體。駱駝和鯊魚除了有正常的 IgG 抗體外,還有一種小抗體,是只有 heavy chain 的抗體。這種小抗體有跟正常抗體一樣的功能,同樣能抓住抗原(antigen),是免疫系統的一部份,在外來物體入侵時,體內會產生免疫反應製造出這些小抗體。小抗體的 variable region,也就是 VHH,又稱 nanobodies,近年被廣泛研究,看是否能用在治療疾病上面。Nanobody 的其中一種用法就是在它的一端接上讓蛋白降解訊息片段(signal sequence),例如 degron,這樣當 nanobody 抓到致病蛋白後,接著就會被帶去 proteasome 或是 lysosome 進行降解。
相關舊文:降解蛋白質的密碼(degron)
小抗體的發現是個意外,VUB 的學生想要分析血液和疾病的關聯的時候,因為不想殺老鼠取血,便想到了冷凍庫有駱駝寫的庫存,拿來用後卻發現駱駝血裡面除了有正常的 IgG,還有一種小型的抗體,研究後發現是沒有 light chain 的抗體。之後英國的博士生想要研究駱駝的小抗體,便請 VUB 的研究人員寄血液檢體砍他,沒想到在運送過程中血液莫名弄丟了。不過呢,之後他發現鯊魚血液裡也有這種只有 heavy chain 的小抗體。(這真是太神奇了,為什麼研究會從駱駝跳到鯊魚呢?)
Nanobody 也讓 Kobeilka 得到 2012 年的諾貝爾化學獎,Kobeilka 研究的是 β2-adrenergic receptor,他利用 nanobodies 把 receptor 鎖在 active state 以研究其 crystal structure,他把結果發表在 2011 年的 Nature,之後便在 2012 年因這項研究得到諾貝爾獎。
References:
Science / Mini-antibodies discovered in sharks and camels could lead to drugs for cancer and other diseases (May 2018)
S. Steeland et al, Nanobodies as therapeutics: big opportunities for small antibodies. Drug Discovery Today (2016)
P. Holliger & P.J. Hudson, Engineered antibody fragments and the rise of single domains. Nature Biotechnology (2005)
Rasmussen et al, Structure of a nanobody-stabilized active state of the β2 adrenoceptor. Nature (2011)
2018年1月14日 星期日
怎樣才算 hard working
對不是我們這個領域的人來說,週末還要到實驗室叫做 hard working,但週末到實驗室其實沒什麼,尤其是如果只是禮拜六來收個實驗,或是禮拜天來用個 inoculation 好隔天可以做 miniprep。真要我週末來待整天,我還無法,會覺得好累,感覺沒放到假,對我就是草莓,我一直都覺得我算是很偷懶的 slacker,尤其跟我們實驗室的中國交換生相比更是(來很久變加拿大人的中國人週末幾乎不會來,除了我老闆),他們都是週末兩天來一天半的(沒來的半天是因為要買菜),像他們週末還跑 Western 這種事我是不會做的,除非真的有必要(不過到目前為止還沒有)。
但我之前被每天都待到七、八點,週末還都在實驗室待整天的中國人說我真是 hard working 時真是不解我 hard working 在哪?他是說他都快中午才來,他來的時候我做事都做到一半了,但是他待到八、九點,我都六點就走人了啊,而且我週末最多只待半天,除非我真的需要一個人靜一靜才會待比較久。
說到這,想到我研究所時系上同學週末也都是會來的,但也是只待個半天,就是收個實驗或做些簡單的實驗而已。然後從某天開始,隔壁實驗室的兩位同學說,他們覺得週末來實驗並不會做得比較順,做不出來的還是做不出來,並沒有因為週末來就做出來了,做得出來的週末沒來還是做得出來。他們經過比較之後,發現週末有來跟沒來結果並沒有差,實驗進度還是一樣,並沒有變得比較快或比較順,那週末幹嘛來呢?所以之後他們週末就不來了。
又想到我在大學時,在實驗室幫一位博士生做他的 side project,他本來都是早上八、九就點來的,但是從某天開始十點多才來,原因是他發現,他早來是因為想早走,但結果還是待到七、八點才走,那幹嘛早來呢?於是他就改成十點多才進實驗室。
之前 UBC 校長也說,研究生活不用只有工作,還是要有正常人的生活,週末多去交朋友,有些私人時間,多些社交空間,work-life balance 很重要,這也是他的親身經驗,他在研究不順的時候讓自己放鬆一下,過回正常人的生活後,研究就變順了。
好了,其實這篇只是想突然有感而發,覺得其實生活還是很重要的,還是需要 work life balance 才不會 burnout。
#加拿大人真沒競爭力XD
但我之前被每天都待到七、八點,週末還都在實驗室待整天的中國人說我真是 hard working 時真是不解我 hard working 在哪?他是說他都快中午才來,他來的時候我做事都做到一半了,但是他待到八、九點,我都六點就走人了啊,而且我週末最多只待半天,除非我真的需要一個人靜一靜才會待比較久。
說到這,想到我研究所時系上同學週末也都是會來的,但也是只待個半天,就是收個實驗或做些簡單的實驗而已。然後從某天開始,隔壁實驗室的兩位同學說,他們覺得週末來實驗並不會做得比較順,做不出來的還是做不出來,並沒有因為週末來就做出來了,做得出來的週末沒來還是做得出來。他們經過比較之後,發現週末有來跟沒來結果並沒有差,實驗進度還是一樣,並沒有變得比較快或比較順,那週末幹嘛來呢?所以之後他們週末就不來了。
又想到我在大學時,在實驗室幫一位博士生做他的 side project,他本來都是早上八、九就點來的,但是從某天開始十點多才來,原因是他發現,他早來是因為想早走,但結果還是待到七、八點才走,那幹嘛早來呢?於是他就改成十點多才進實驗室。
之前 UBC 校長也說,研究生活不用只有工作,還是要有正常人的生活,週末多去交朋友,有些私人時間,多些社交空間,work-life balance 很重要,這也是他的親身經驗,他在研究不順的時候讓自己放鬆一下,過回正常人的生活後,研究就變順了。
好了,其實這篇只是想突然有感而發,覺得其實生活還是很重要的,還是需要 work life balance 才不會 burnout。
#加拿大人真沒競爭力XD
2018年1月13日 星期六
利用 ASO 治療阿茲海默症
阿茲海默症(Alzheimer's Disease, AD)的致病原因除了大家都知道的 β-amyloid (Aβ) 和 tau 堆積外,還有一個是 ApoE (apolipoprotein E)。ApoE 在腦中是由 astrocytes 製造的,原本的功能是運送膽固醇(cholesterol)到神經細胞,但並不知道它在 AD 裡扮演的角色為何。雖然早在 25 年前就發現它會增加 AD 的風險,帶有 ApoE4 基因得到 AD 的機率比帶有 ApoE3 的高三倍以上,但其詳細機制還不清楚。
目前認為 Aβ 是引起阿茲海默症的原因,但它的堆積似乎和疾病的症狀沒有直接關係,在它之後發生的 tau 堆積反而會造成腦部的損害。那 ApoE 呢?它在其中扮演獨立的角色,還是牽線的角色?ApoE 的 variants 有三種:ApoE2-4,並不是每種都會增加阿茲海默症的風險,目前所知只有 ApoE4 會。這期的 Neuron 有兩篇是關於 ApoE4,第一篇研究顯示 ApoE4 是造成 Aβ 開始堆積的原因。之前有研究顯示帶有 ApoE4 基因的人其 Aβ 堆積的 plaques 比沒帶得多,也許它是造成 Aβ 堆積的原因,或是它會促進 Aβ 的堆積。Aβ 在腦中一開始只是緩慢的微量堆積,稱為 seeding stage,到後期量邊多了以後,堆積速度會變快,而在這個過程中 ApoE 是從哪個階段開始參與呢?
Liu et al 的這篇研究用了可以控制 ApoE3 和 ApoE4 表現且帶有基因突變(APP/PS1)的 AD 老鼠,AD 老鼠在四、五個月大的時候會開始出現 Aβ 堆積的徵兆,然後六到九個月大是 Aβ 堆積最快速的時期,十個月大的時候則接近巔峰。他們讓老鼠的 ApoE 在三個不同時期開始表現:從剛出生到九個月大時都持續表現、只在前面六個月(也就是 Aβ 的 seeding stage)表現,或是在疾病的後期(也就是六到九個月大時)才表現。他們發現 ApoE4 從剛出生到九個月大都持續表現的老鼠,牠們的 Aβ 堆積有明顯的增加,而只表現 ApoE3 的老鼠則沒出現這個現象。他們也比較了只表現前六個月(出生到六個月大)和只表現後三個月(六到九個月大)的老鼠,發現前六個月有 ApoE4 表現的老鼠的 Aβ 堆積也有明顯增加,而只有後三個月有 ApoE4 表現的老鼠卻沒這個現象。另外,他們也發現 ApoE4 會讓 Aβ 無法被自然清理掉,使得它更容易堆積在腦內。
ApoE4 除了會加速 Aβ 的堆積外,也會增加 tau 對腦部的傷害。幾個月前刊在 Nature 的研究顯示 ApoE4 會加速 tau 的堆積,之前也有研究顯示 ApoE4 會增加 phospho-tau (p-tau) 的表現量。 Shi et al 的這個研究用了帶有突變 tau 基因(P301S)的老鼠,這個突變會產生大量 tau (是原本 endogenous tau 的五倍量),隨著年紀增長,insolube tau 會漸漸增加,最後形成 tau tangles,且這些堆積在腦部的 tau 都被高度磷酸化(phosphorylated)。這種老鼠在 1.5 個月大的時候腦部就會出現 tau 的堆積,在八個月大的時候即可觀察到腦細胞死亡和腦萎縮,海馬迴尤為嚴重。他們把這種老鼠本身的 ApoE4 基因拿掉,讓它不帶任何 ApoE 基因(ApoE KO, TEKO),或是拿掉 ApoE4 基因(TEKO)後再轉進其中一種人類的 ApoE 基因(P301S/ApoE2 (TE2), P301S/ApoE3 (TE3), P301S/ApoE4 (TE4))。
About AD transgenic mice: Tau P301S (Line PS19)
突變 tau 的老鼠在三個月大時,帶有人類 ApoE4 基因的老鼠腦部的 tau 表現量就比帶有其他種 ApoE 的高很多,其海馬迴裡的 p-tau 表現量也比較高。當老鼠九個月大的時候,他們檢視老鼠腦部,發現帶有 ApoE4 基因的老鼠腦部損害(萎縮)最嚴重,而帶有 ApoE2 的則是最輕。有趣的是沒帶有任何 ApoE 基因的老鼠(P301S/TEKO)腦部幾乎沒有被 tau tangles 損害;而不管是否帶有其中一種 ApoE,但是只帶有正常 tau 而非突變 tau 基因的老鼠,在九個月大的時候腦部也沒有萎縮的跡象。另外,帶有 ApoE4 基因的老鼠 (P301S/TE4)出現因為免疫反應引起的發炎,但是帶有其他 ApoE 的老鼠並沒有這個情形。而且同樣的,如果是正常 tau 而非突變 tau 的話,即便帶有 ApoE 也沒有出現免疫反應。體外試驗顯示並非只有 ApoE4 會使表現突變 tau 的細胞出現免疫反應和引起神經細胞的死亡,其他的 ApoE 也會,只是沒 ApoE4 那麼嚴重。
除了老鼠實驗,他們也檢視了 ApoE 和 tau 在人類患者中的關係是否和在老鼠中相同。大多數人帶有的是 ApoE3,少數沒有帶任何 ApoE 基因的人通常會出現膽固醇過高的情形,如果沒治療的話,可能會在年輕時便因心血管疾病而死。在這個研究裡, 他們檢視了 79 位因為 tau 堆積致死患者的大腦和他們帶有的 ApoE 基因,發現 Aβ 堆積的程度和腦部萎縮的程度沒有直接關係,反倒是帶有 ApoE4 的患者惡化的比較快,腦部萎縮、損害的比較嚴重。總結來說,這篇研究顯示相較於 Aβ,tau 和 ApoE4 比較像是使病情惡化的主因,ApoE 會加速 p-tau 的堆積,並且引發免疫反應造成發炎現象,最終導致腦細胞死亡。

既然 ApoE4 似乎是造成疾病的關鍵因素之一,於是這期 Neuron 的第二篇相關研究便是用了ASO (antisense oligo)去抑制 ApoE 的表現,看看是否會有效果。他們用了兩種老鼠,分別是帶有 AD 突變基因(APP/PS1)和人類 ApoE3 的老鼠(AD/ApoE3)和帶有人類 ApoE4 的 AD 老鼠(AD/ApoE4),然後在不同的時期把 ASO 打入這兩種老鼠:從剛出生第一天就給 ASO,或從六週大時(腦中已出現 plaques)才給 ASO,然後在十六週(四個月)大的時候檢視這兩種老鼠腦中的 Aβ 堆積情形。結果發現不管從何時開始打 ASO 都能有效降低 ApoE3 和 ApoE4 至約一半的量。至於在 Aβ 的部分,剛出生就給與 ASO 治療的老鼠,不管是 ApoE3 還是 ApoE4 的老鼠,牠們腦中的 Aβ 都明顯的降低了,並且 ApoE4 老鼠腦中的 plaques 也比沒 ASO 治療的低。如果是六週後才進行 ASO 治療的話,Aβ 量則沒有明顯的降低。不過有趣的是不管是剛出生就給 ASO,還是六週後才給 ASO,Aβ plaques 旁邊的 neuritic dystrophy 都有變少,即便六週才給 ASO 的老鼠腦中的 Aβ 量並沒有減少,表示 ApoE4 可能有參與 Aβ 引起的發炎反應。
由以上研究看來,ApoE4 雖然是配角,但卻是關鍵配角。好想知道我的 ApoE variants 是哪一種哦,希望不是 ApoE4。XD
延伸閱讀:阿茲海默症新藥 Aducanumab
Articles:
WU in St. Louis Press Release / Newly ID’d role of major Alzheimer’s gene suggests possible therapeutic target
Alzheimer's News Today / Targeting ApoE in Brain Alone May Lead to Alzheimer’s Treatment, Researchers Say
NNR / Treatment Reduces Alzheimer's Damage
Nature News / Alzheimer's disease: The forgetting gene (2014)
Papers:
Y Yoshiyama et al, Synapse Loss and Microglial Activation Precede Tangles in a P301S Tauopathy Mouse Model. Neuron (2007)
Y Shi et al, ApoE4 markedly exacerbates tau-mediated neurodegeneration in a mouse model of tauopathy. Nature (2017)
C Liu et al, ApoE4 Accelerates Early Seeding of Amyloid Pathology. Neuron (2017)
TV Huynh et al, Age-Dependent Effects of apoE Reduction Using Antisense Oligonucleotides in a Model of β-amyloidosis. Neuron (2017)
目前認為 Aβ 是引起阿茲海默症的原因,但它的堆積似乎和疾病的症狀沒有直接關係,在它之後發生的 tau 堆積反而會造成腦部的損害。那 ApoE 呢?它在其中扮演獨立的角色,還是牽線的角色?ApoE 的 variants 有三種:ApoE2-4,並不是每種都會增加阿茲海默症的風險,目前所知只有 ApoE4 會。這期的 Neuron 有兩篇是關於 ApoE4,第一篇研究顯示 ApoE4 是造成 Aβ 開始堆積的原因。之前有研究顯示帶有 ApoE4 基因的人其 Aβ 堆積的 plaques 比沒帶得多,也許它是造成 Aβ 堆積的原因,或是它會促進 Aβ 的堆積。Aβ 在腦中一開始只是緩慢的微量堆積,稱為 seeding stage,到後期量邊多了以後,堆積速度會變快,而在這個過程中 ApoE 是從哪個階段開始參與呢?
Liu et al 的這篇研究用了可以控制 ApoE3 和 ApoE4 表現且帶有基因突變(APP/PS1)的 AD 老鼠,AD 老鼠在四、五個月大的時候會開始出現 Aβ 堆積的徵兆,然後六到九個月大是 Aβ 堆積最快速的時期,十個月大的時候則接近巔峰。他們讓老鼠的 ApoE 在三個不同時期開始表現:從剛出生到九個月大時都持續表現、只在前面六個月(也就是 Aβ 的 seeding stage)表現,或是在疾病的後期(也就是六到九個月大時)才表現。他們發現 ApoE4 從剛出生到九個月大都持續表現的老鼠,牠們的 Aβ 堆積有明顯的增加,而只表現 ApoE3 的老鼠則沒出現這個現象。他們也比較了只表現前六個月(出生到六個月大)和只表現後三個月(六到九個月大)的老鼠,發現前六個月有 ApoE4 表現的老鼠的 Aβ 堆積也有明顯增加,而只有後三個月有 ApoE4 表現的老鼠卻沒這個現象。另外,他們也發現 ApoE4 會讓 Aβ 無法被自然清理掉,使得它更容易堆積在腦內。
ApoE4 除了會加速 Aβ 的堆積外,也會增加 tau 對腦部的傷害。幾個月前刊在 Nature 的研究顯示 ApoE4 會加速 tau 的堆積,之前也有研究顯示 ApoE4 會增加 phospho-tau (p-tau) 的表現量。 Shi et al 的這個研究用了帶有突變 tau 基因(P301S)的老鼠,這個突變會產生大量 tau (是原本 endogenous tau 的五倍量),隨著年紀增長,insolube tau 會漸漸增加,最後形成 tau tangles,且這些堆積在腦部的 tau 都被高度磷酸化(phosphorylated)。這種老鼠在 1.5 個月大的時候腦部就會出現 tau 的堆積,在八個月大的時候即可觀察到腦細胞死亡和腦萎縮,海馬迴尤為嚴重。他們把這種老鼠本身的 ApoE4 基因拿掉,讓它不帶任何 ApoE 基因(ApoE KO, TEKO),或是拿掉 ApoE4 基因(TEKO)後再轉進其中一種人類的 ApoE 基因(P301S/ApoE2 (TE2), P301S/ApoE3 (TE3), P301S/ApoE4 (TE4))。
About AD transgenic mice: Tau P301S (Line PS19)
突變 tau 的老鼠在三個月大時,帶有人類 ApoE4 基因的老鼠腦部的 tau 表現量就比帶有其他種 ApoE 的高很多,其海馬迴裡的 p-tau 表現量也比較高。當老鼠九個月大的時候,他們檢視老鼠腦部,發現帶有 ApoE4 基因的老鼠腦部損害(萎縮)最嚴重,而帶有 ApoE2 的則是最輕。有趣的是沒帶有任何 ApoE 基因的老鼠(P301S/TEKO)腦部幾乎沒有被 tau tangles 損害;而不管是否帶有其中一種 ApoE,但是只帶有正常 tau 而非突變 tau 基因的老鼠,在九個月大的時候腦部也沒有萎縮的跡象。另外,帶有 ApoE4 基因的老鼠 (P301S/TE4)出現因為免疫反應引起的發炎,但是帶有其他 ApoE 的老鼠並沒有這個情形。而且同樣的,如果是正常 tau 而非突變 tau 的話,即便帶有 ApoE 也沒有出現免疫反應。體外試驗顯示並非只有 ApoE4 會使表現突變 tau 的細胞出現免疫反應和引起神經細胞的死亡,其他的 ApoE 也會,只是沒 ApoE4 那麼嚴重。
除了老鼠實驗,他們也檢視了 ApoE 和 tau 在人類患者中的關係是否和在老鼠中相同。大多數人帶有的是 ApoE3,少數沒有帶任何 ApoE 基因的人通常會出現膽固醇過高的情形,如果沒治療的話,可能會在年輕時便因心血管疾病而死。在這個研究裡, 他們檢視了 79 位因為 tau 堆積致死患者的大腦和他們帶有的 ApoE 基因,發現 Aβ 堆積的程度和腦部萎縮的程度沒有直接關係,反倒是帶有 ApoE4 的患者惡化的比較快,腦部萎縮、損害的比較嚴重。總結來說,這篇研究顯示相較於 Aβ,tau 和 ApoE4 比較像是使病情惡化的主因,ApoE 會加速 p-tau 的堆積,並且引發免疫反應造成發炎現象,最終導致腦細胞死亡。

既然 ApoE4 似乎是造成疾病的關鍵因素之一,於是這期 Neuron 的第二篇相關研究便是用了ASO (antisense oligo)去抑制 ApoE 的表現,看看是否會有效果。他們用了兩種老鼠,分別是帶有 AD 突變基因(APP/PS1)和人類 ApoE3 的老鼠(AD/ApoE3)和帶有人類 ApoE4 的 AD 老鼠(AD/ApoE4),然後在不同的時期把 ASO 打入這兩種老鼠:從剛出生第一天就給 ASO,或從六週大時(腦中已出現 plaques)才給 ASO,然後在十六週(四個月)大的時候檢視這兩種老鼠腦中的 Aβ 堆積情形。結果發現不管從何時開始打 ASO 都能有效降低 ApoE3 和 ApoE4 至約一半的量。至於在 Aβ 的部分,剛出生就給與 ASO 治療的老鼠,不管是 ApoE3 還是 ApoE4 的老鼠,牠們腦中的 Aβ 都明顯的降低了,並且 ApoE4 老鼠腦中的 plaques 也比沒 ASO 治療的低。如果是六週後才進行 ASO 治療的話,Aβ 量則沒有明顯的降低。不過有趣的是不管是剛出生就給 ASO,還是六週後才給 ASO,Aβ plaques 旁邊的 neuritic dystrophy 都有變少,即便六週才給 ASO 的老鼠腦中的 Aβ 量並沒有減少,表示 ApoE4 可能有參與 Aβ 引起的發炎反應。
由以上研究看來,ApoE4 雖然是配角,但卻是關鍵配角。好想知道我的 ApoE variants 是哪一種哦,希望不是 ApoE4。XD
延伸閱讀:阿茲海默症新藥 Aducanumab
Articles:
WU in St. Louis Press Release / Newly ID’d role of major Alzheimer’s gene suggests possible therapeutic target
Alzheimer's News Today / Targeting ApoE in Brain Alone May Lead to Alzheimer’s Treatment, Researchers Say
NNR / Treatment Reduces Alzheimer's Damage
Nature News / Alzheimer's disease: The forgetting gene (2014)
Papers:
Y Yoshiyama et al, Synapse Loss and Microglial Activation Precede Tangles in a P301S Tauopathy Mouse Model. Neuron (2007)
Y Shi et al, ApoE4 markedly exacerbates tau-mediated neurodegeneration in a mouse model of tauopathy. Nature (2017)
C Liu et al, ApoE4 Accelerates Early Seeding of Amyloid Pathology. Neuron (2017)
TV Huynh et al, Age-Dependent Effects of apoE Reduction Using Antisense Oligonucleotides in a Model of β-amyloidosis. Neuron (2017)
2018年1月10日 星期三
你的 strengths 是什麼?
今天去參加了一個 post-doc office 辦的 career workshop,在去之前他會要大家先上 Gallup Strength 做測試,看你的 strengths 在哪裡,因為有個理論是說,與其過份在意你的 weakness,不如好好了解你的 strengths,然後充份運用它、加強它,然後好好利用自己的 strengths,比較會在工作得到成就感和享受工作。另外有個比較有名的是 MBTI tests (費用也貴很多),也是測試人格測驗的一種,有的公司會付費讓員工做,我妹之前的公司就有讓他們做,加拿大政府好像也有讓政府員工做,主要是讓員工能更認識自己的特質,更能有效得工作。Gallup 檢測系統歸類了 34 種 strengths,學校付費讓我們免費做測試(原價好像是 CAD$25),可以得出你的 top 5 strengths (如果你想知道更多,例如你的全部 34 種的排列的話,則要加錢)。
Workshop 舉辦者有把所有參與者的 strengths 整理出來(如圖),看每個 stregnth 有多少人有,有趣的是你可以看到大多數人的 strengths 都集中在兩端:executing 和 strategic thinking。有趣的點在哪?去參加這個 workshop 的都是博士後,大家的 strengths 好像很理所當然都集中在這兩樣,似乎因為有了這些特質或 strengths 所以才有辦法在博班生涯中生存下來 XD,其中有一個人的 top 5 都是紅色的,聽主講人說這種人非常罕見(但我發現其實有好幾個人有四個 strengths 都是紅色的)。另一個有趣的點是最少的是 influencing 的部分,relationship building 可能因為研究或多或少需要合作,所以這部分還是有,但跟 executing 和 strategic thinking 比起來還是少很多。

這個統計反映出了一個 .... 算是教育系統還是人才培養系統需要加強的部分嗎?你會覺得這些人應該是要對社會有某些影響力的,但是他們(包括我)最欠缺的卻是這方面的能力,當然也許不是欠缺,畢竟這只是 top 5 的分析而已,可能是相較於其他 strengths,這些是比較弱的部分。如果讓商學院或社會學的來做這些,可能大家的 strengths 都集中在中間那兩項吧。XD
Executing: Leaders with dominant strength in the Executing domain know how to make things happen. When you need someone to implement a solution, these are people who will work tireless to get it done. Leaders with a strength to execute have the ability to "catch" an idea and make it reality.
Influencing: Those who lead by influencing help their team reach a much broader audience. People with strength in this domain are always selling the team's idea inside and outside the organization. When you need someone to take charge, speak up, and make sure your group is heard, look to someone the the strength to influence.
Relationship Building: Those who lead through Relationship Building are the essential glue that holds a team together. Without these strengths on a team, in many cases, the group is simply a composite of individuals. In contrast, leaders with exceptional Relationship Building strength have the unique ability to create groups and organizations that are much greater than the sum of their parts.
Strategic Thinking: Leaders with great strategic Thinking are the ones who keep us all focused on could be. They are constantly absorbing and analyzing information and helping the team make better decisions. People with strength in this strength continually stretch our thinking for the future.
Workshop 舉辦者有把所有參與者的 strengths 整理出來(如圖),看每個 stregnth 有多少人有,有趣的是你可以看到大多數人的 strengths 都集中在兩端:executing 和 strategic thinking。有趣的點在哪?去參加這個 workshop 的都是博士後,大家的 strengths 好像很理所當然都集中在這兩樣,似乎因為有了這些特質或 strengths 所以才有辦法在博班生涯中生存下來 XD,其中有一個人的 top 5 都是紅色的,聽主講人說這種人非常罕見(但我發現其實有好幾個人有四個 strengths 都是紅色的)。另一個有趣的點是最少的是 influencing 的部分,relationship building 可能因為研究或多或少需要合作,所以這部分還是有,但跟 executing 和 strategic thinking 比起來還是少很多。

這個統計反映出了一個 .... 算是教育系統還是人才培養系統需要加強的部分嗎?你會覺得這些人應該是要對社會有某些影響力的,但是他們(包括我)最欠缺的卻是這方面的能力,當然也許不是欠缺,畢竟這只是 top 5 的分析而已,可能是相較於其他 strengths,這些是比較弱的部分。如果讓商學院或社會學的來做這些,可能大家的 strengths 都集中在中間那兩項吧。XD
Executing: Leaders with dominant strength in the Executing domain know how to make things happen. When you need someone to implement a solution, these are people who will work tireless to get it done. Leaders with a strength to execute have the ability to "catch" an idea and make it reality.
Influencing: Those who lead by influencing help their team reach a much broader audience. People with strength in this domain are always selling the team's idea inside and outside the organization. When you need someone to take charge, speak up, and make sure your group is heard, look to someone the the strength to influence.
Relationship Building: Those who lead through Relationship Building are the essential glue that holds a team together. Without these strengths on a team, in many cases, the group is simply a composite of individuals. In contrast, leaders with exceptional Relationship Building strength have the unique ability to create groups and organizations that are much greater than the sum of their parts.
Strategic Thinking: Leaders with great strategic Thinking are the ones who keep us all focused on could be. They are constantly absorbing and analyzing information and helping the team make better decisions. People with strength in this strength continually stretch our thinking for the future.
2018年1月8日 星期一
腸道菌也許可以用來治療氣喘和過敏
下面列點筆記,沒有特別整理。
* 太乾淨的環境可能誤導免疫系統,使它在遇到些微的外來物(包括食物中的蛋白質)時變反應太過,造成過敏。
* 嬰兒在一歲就接受抗生素,得到氣喘的比例比較高。
* 他們給 germ-free mice (生長在無菌環境裡的無菌鼠)幼鼠和成鼠抗生素時,之後再給牠們常用的過敏原 ovalbumin (OVA) 去引發過敏反應,結果只有幼鼠出現明顯的哮喘反應,抗生素對成鼠沒有產生哮喘反應。另外,抗生素也影響了幼鼠和成鼠的腸道菌落,但是不同的改變。
* 他們收集了三百多個嬰兒出生時、三個月大時和一歲時的糞便,他們檢驗分析後,發現有四種腸道菌在有氣喘和沒氣喘的小孩中是不同的:Faecalibacterium, Lachnospira, Veillonella & Rothia。他們把這四種集合稱為 FLVR,音為 flavor。如果小孩在三個月大時有這四種細菌,得到氣喘的機率就很小。反之,如果你的 FLVR 很少,那就有較高的機率有氣喘。之後,他們把 FLVR 給無菌鼠吃,吃了細菌的無菌鼠出現肺炎和氣喘相關症狀比沒吃的還要輕。
* 去年 UCSF 的一篇研究顯示,當嬰兒在一個月大時如果缺少某些腸道菌,那他們在兩歲時出現過敏症狀和在四歲時出現氣喘症狀的機率比其他人高三倍。
* 2014 年時有篇研究顯示,五天的健康飲食就可以改變腸道菌的生態,吃很多青菜(plant-based diet)的人的腸道菌比吃很多肉(meat-based diet)的人健康。
* 家裡有養狗或貓的人,得到過敏或氣喘的機率比較小。家裡有養寵物的人,體內腸道菌落的種類比較多。
* 體內的腸道菌落是互相依附和互相影響的,因此產生出獨特的環境,改變一種細菌就會改變整個環境。
* 腸道菌並不是你吃進去後就會順利在你體內住下來了,很多情況是過幾天後它們就消失了,所以需要每天吃。
Articles:
Allergic Living / Why Are There So Many Food Allergies Today? (by Dr. S. Sicherer) Allergic Living (2015)
CIFAR / Missing bacteria linked with asthma (2015)
UCSC / Interview: B. Brett Finlay, microbiologist (2017)
Allergic Living / Inside the Microbiome: Why Good Gut Bacteria Is the Big Hope For Allergic Disease. (2017)
Papers:
SL Russell et al, Early life antibiotic‐driven changes in microbiota enhance susceptibility to allergic asthma. EMBO Reports (2012)
LA David et al, Diet rapidly and reproducibly alters the human gut microbiome. Nature (2014)
M Arrieta et al, Early infancy microbial and metabolic alterations affect risk of childhood asthma. Science Translational Medicine (2015)
* 太乾淨的環境可能誤導免疫系統,使它在遇到些微的外來物(包括食物中的蛋白質)時變反應太過,造成過敏。
* 嬰兒在一歲就接受抗生素,得到氣喘的比例比較高。
* 他們給 germ-free mice (生長在無菌環境裡的無菌鼠)幼鼠和成鼠抗生素時,之後再給牠們常用的過敏原 ovalbumin (OVA) 去引發過敏反應,結果只有幼鼠出現明顯的哮喘反應,抗生素對成鼠沒有產生哮喘反應。另外,抗生素也影響了幼鼠和成鼠的腸道菌落,但是不同的改變。
* 他們收集了三百多個嬰兒出生時、三個月大時和一歲時的糞便,他們檢驗分析後,發現有四種腸道菌在有氣喘和沒氣喘的小孩中是不同的:Faecalibacterium, Lachnospira, Veillonella & Rothia。他們把這四種集合稱為 FLVR,音為 flavor。如果小孩在三個月大時有這四種細菌,得到氣喘的機率就很小。反之,如果你的 FLVR 很少,那就有較高的機率有氣喘。之後,他們把 FLVR 給無菌鼠吃,吃了細菌的無菌鼠出現肺炎和氣喘相關症狀比沒吃的還要輕。
* 去年 UCSF 的一篇研究顯示,當嬰兒在一個月大時如果缺少某些腸道菌,那他們在兩歲時出現過敏症狀和在四歲時出現氣喘症狀的機率比其他人高三倍。
* 2014 年時有篇研究顯示,五天的健康飲食就可以改變腸道菌的生態,吃很多青菜(plant-based diet)的人的腸道菌比吃很多肉(meat-based diet)的人健康。
* 家裡有養狗或貓的人,得到過敏或氣喘的機率比較小。家裡有養寵物的人,體內腸道菌落的種類比較多。
* 體內的腸道菌落是互相依附和互相影響的,因此產生出獨特的環境,改變一種細菌就會改變整個環境。
* 腸道菌並不是你吃進去後就會順利在你體內住下來了,很多情況是過幾天後它們就消失了,所以需要每天吃。
Articles:
Allergic Living / Why Are There So Many Food Allergies Today? (by Dr. S. Sicherer) Allergic Living (2015)
CIFAR / Missing bacteria linked with asthma (2015)
UCSC / Interview: B. Brett Finlay, microbiologist (2017)
Allergic Living / Inside the Microbiome: Why Good Gut Bacteria Is the Big Hope For Allergic Disease. (2017)
Papers:
SL Russell et al, Early life antibiotic‐driven changes in microbiota enhance susceptibility to allergic asthma. EMBO Reports (2012)
LA David et al, Diet rapidly and reproducibly alters the human gut microbiome. Nature (2014)
M Arrieta et al, Early infancy microbial and metabolic alterations affect risk of childhood asthma. Science Translational Medicine (2015)
2017年12月23日 星期六
R | Data manipulation (2): arrange, mutate, summarise
這篇介紹另外幾個處理資料的功能,上篇請看這裡:R | Data manipulation (1): select, filter, slice
詳細功能介紹請參考這兩頁:Working with Data - Part 1 & Intro to R - Part 3
練習的部分可以參考 slides: Working with Data - Part 1 (pdf)
這幾個功能在 dplyr 這個 package 裡面,所以要先跑。同時要用內建檔案 mtcars 來練習,所以也要叫出來。下面還會用檔案資料 flights,因為它在 nycflights13 這個 packages 裡面,所以也需要安裝。
下面先介紹一個觀看檔案的功能。
Glimpse function: columns run down the page, and data runs across, making it possible to see every column in a data frame
glimpse() 和平常看檔案的方式不一樣。我們平常看檔案的時候,variables 是在第一列(rows),每個欄位(column)是一個 variables。但用 glimpse() 的話,它會橫向顯示每個 variable 的數據值。
下面先用資料檔案 flights 練習。
dim: 顯示檔案資料的大小 dimension (row x column)
上面顯示的結果告訴我們,共有 336776 個班機(observations),且用 19 個 variables 呈現各個班機的資訊。
上面用 glimpse() 功能可以看到每班飛機的資訊(variables)包括有 year, month, day, dep_time 等等。
5. Arrange function: order observations (rows)
沒特別指令的話會由小到大排列。
Default setting: order mpg values from small to large. (依 mpg 的大小排列)
如果要由大到小排列的話,則用:desc()
Use desc() to sort in descending order.
Order mpg from large to small.
如果有兩個 variables 的話,會先排第一個,然後再在第一個裡面排第二個的大小順序。
Arrange with cyl first; then within cyl, arrange gear.
上面的例子中,會先依照 cyl 的大小排列,再依 gear 的大小排列。
如果想讓兩個 variables 都由大到小排列,可以分開寫,也可以合在一起。
Reorder the mtcars in descending order of mpg and displacement (disp)
或寫成這樣:
從上面可以看到是先依 carb 的大小排列,之後再依 mpg 的大小排列(最後四排當 carb 都是 4 的時候,mpg 是由大到小排列),但兩個都是由大到小。
Exercise
這邊我們用檔案資料 flights 來練習。
Q1. Which flights are the most delayed?
哪個班機起飛時間延遲最多,可以用 desc() 由大到小排列延遲的長度,第一個就是延遲最多的。下面先用 glimpse 呈現結果。
從上面顯示的最後一行 dep_delay 可以看到是由大到小排列。
如果不確定哪個 variable 是指延遲時間的話,可以用 ? 或 help() 的功能來查看:?flights 或是 help(flights)。
會在 R Studio 右下角的視窗中出現解釋框(如下圖,點圖可以放大),裡面寫:
下面是沒用 glimpse() 功能呈現,應該會比較清楚。
Q2: Which flights caught up the most time during the flight?
哪班飛機趕上的時間最多,就是說即便是延遲起飛,但是仍然準時抵達的,也就是延遲時間最短的(比預計的飛行時間短),這邊用延遲起飛的時間減掉延遲抵達的時間,再由大到小排列。
不用 glimpse 顯示:
可以指定 catchup 為 flights,再帶入 arrange,或是用 head 只顯示前十個 carrier。
如果只看資料的前十個的話,則是下面這樣。
6. Mutate function: 設定新的 variable,新設定的 variable 會顯示在最後一欄。
Make new variables: disp_l and wt_kg
1L = 61.0237 cu.in. (cubic inch)
1kg = 2.2 lbs
檔案資料裡面的 disp 單位是 cu.in.,我們把它轉換成 L,設定其為 disp_l。同時也把裡面原本單位為磅(lbs)的 wt 換算成 kg,指定其為 wt_kg。新訂的 disp_l 和 wt_kg 會顯示在最後兩的 column。
把重量換算成噸,指定其為:wt_tonnes
Include a variable for weight in tonnes (1t = 2,204.6 lbs)
transmute: leave only the new variables
如果用 transmute() 的話,就只會顯示新訂的 disp_1 和 wt_kg。
Exercise
Q1. Compute speed in mph from time (in minutes) and distance (in miles)
同樣可以用 ?flights 查詢:
計算飛機的速度:mph = miles per hour
也就是飛行的距離 distance (in miles) 除以飛行的時間 air_time (in minutes)。
因為 air_time 是以分鐘為單位,所以我們必須先把它轉換成小時,也就是:air_time / 60
下面把速度指定為 speed。
顯示整個檔案太長,看不到後面計算出來的 speed,所以用 select() 的功能挑出我們想看的幾個 variables。如果忘記 select() 是什麼、怎麼用的話,可以看前一篇。
Q2. Which flight flew the fastest?
哪個班機的速度最快?可以用 arrange() 功能由大到小排列出來,跟上面一樣只挑出我們想看的幾個 variables 來看。
Q3. 把上面班機延遲練習的 Q2,用 select() 的功能挑出我們想看的幾項出來。
可以和上面排列前十的 carrier 的結果比對,看排列是不是一樣的。
7. Summarise / Summarize
功能和 mutate() 有點像,不一樣的是它會產生一個新的 data frame,並且是那欄(也就是那個 variable)所有觀察資料的總結。
例如算出 mpg 的平均值,可以用 mean() 的功能這樣算:
如果用 mutate() 的話會是這樣:
同樣是算出 mean,但它會放在最後一欄,所以所有數值都一樣,因為它是全部觀察算出來的平均值。
如果用 summarise() 的話則是這樣,以一個 data frame 的樣式呈現出來:
也可以幫平均值指定一個名稱 mpg_mean,直接寫在裡面即可。
也可以設定三個,例如把 mpg 的平均值設為 mean_mpg,把重量的中間值設為 median_wt,然後把這兩者的比例設為 ratio。
找出中間值用的功能是:median()
也可以全部寫在一行,下面同時把 mpg 的平均值直接設為 mpg,wt 的中間值設為 wt,把兩者的比例設為 ratio。
除了算全部的平均值外,也可以分組算,例如算出 cyl 裡各種觀察值的平均值。下面先用 levels() 看 cyl 裡面有哪幾種,因為 cyl 的觀察值是數字,需要先把它換成 factor。
由上面結果可以看出 cyl 的觀察值有三種:4, 6, 8。如果要看各個的 mpg 和重量平均值,可以用 group_by() 的功能。
也可以在一組裡面再分組算平均值,例如先用 am 分組後,在再裡面用 cyl 分組,先分的那個放前面,所以語法會是:group_by(am, cyl)
因為用 summarise() 算出來的結果本身就會轉換成一個 data frame,所以可以把這個 data frame 指定一個名稱,例如為 cars_am_cyl,這樣之後想要看的時候便不需要打全部的語法,只要叫出 cars_am_cyl 即可看。
因為 cars_am_cyl 已經是一個 data frame,也可以直接用它來做運算,因為上面設定時已經先用 am 分好組了,所以算平均值的時候會依這個分。
也可以相反設試試看,先用 cyl 分組後再依 am 分組運算,也就是 group_by(cyl, am),然後和上面比較有何不同。
因為上面是先用 cyl 分組,所以接下來用 cars_cyl_am 來做運算的話,會算出 cyl 裡各組的平均值。
Exercise
Q1. Compute the minimum and maximum displacement for each engine type (vs) by transmission type (am)
在各個引擎的種類中依 am 分組後,找出其中的最大值和最小值,因此要先依引擎分類,再在其中依 am 分組,也就是:group_by(vs, am)
最大值和最小值的功能為:max() 和 min()
Q2. Which destinations have the highest average delays?
8. Boxplot
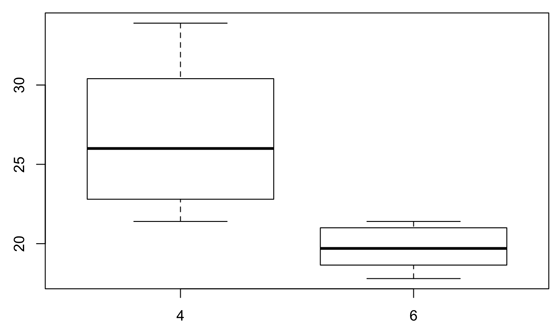
接下來試著畫箱圖,如果想畫出在 cyl < 8 的資料中,mpg 對 cyl 的反應,也就是:
boxplot 的語法是:boxplot(formula, data = )
formula 是指兩軸(也就是兩個 variables)的關係,在這裡是: y ~ x
表示 Y-axis 對 X-axis 的反應,所以也就是:boxplot(y ~ x, data = )
最後用 subset = 的功能挑出你要的,在這個例子裡就是 cyl < 8。
也可以用 %>% 分開寫。
You can use . as a placeholder when the “data” argument is in the second position.
在上面的語法裡,當前面已經表示過用的資料是 mtcars 的時候,後面在 boxplot() 裡面 data 的部分就可以用 "." 替代,也就是:data = .
也可以用 ggplot2 來畫圖,用 ggplot 裡的 geom_boxplot() 畫的話,語法就是這樣:
因為 cyl 是數字,所以要需要先把它變成 factor,想更了解的話可以參考這篇:R | ggplot: Point plot & Box plot
也可以先把畫圖要用的資料用 subset() 的功能挑出來,也就是 cyl < 8 的部分。我們可以把挑出來的部分指定成一個新的 data frame,下面我們把指定其為 cyl_sub,然後再用它來畫圖。

好了,這篇就先到這吧。
詳細功能介紹請參考這兩頁:Working with Data - Part 1 & Intro to R - Part 3
練習的部分可以參考 slides: Working with Data - Part 1 (pdf)
這幾個功能在 dplyr 這個 package 裡面,所以要先跑。同時要用內建檔案 mtcars 來練習,所以也要叫出來。下面還會用檔案資料 flights,因為它在 nycflights13 這個 packages 裡面,所以也需要安裝。
| library(dplyr) library(nycflights13) mtcars flights |
下面先介紹一個觀看檔案的功能。
Glimpse function: columns run down the page, and data runs across, making it possible to see every column in a data frame
glimpse() 和平常看檔案的方式不一樣。我們平常看檔案的時候,variables 是在第一列(rows),每個欄位(column)是一個 variables。但用 glimpse() 的話,它會橫向顯示每個 variable 的數據值。
下面先用資料檔案 flights 練習。
| dim(flights) |
dim: 顯示檔案資料的大小 dimension (row x column)
| [1] 336776 19 |
上面顯示的結果告訴我們,共有 336776 個班機(observations),且用 19 個 variables 呈現各個班機的資訊。
| glimpse(flights) |
|
Observations: 336,776 Variables: 19 $ year <int> 2013, 2013, 2013, 2013, 2013, 2013, ... $ month <int> 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, ... $ day <int> 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, ... $ dep_time <int> 517, 533, 542, 544, 554, 554, 555, ... |
上面用 glimpse() 功能可以看到每班飛機的資訊(variables)包括有 year, month, day, dep_time 等等。
5. Arrange function: order observations (rows)
沒特別指令的話會由小到大排列。
Default setting: order mpg values from small to large. (依 mpg 的大小排列)
| arrange(mtcars, mpg) |
|
mpg cyl disp hp drat wt qsec vs am gear carb 1 10.4 8 472.0 205 2.93 5.250 17.98 0 0 3 4 2 10.4 8 460.0 215 3.00 5.424 17.82 0 0 3 4 3 13.3 8 350.0 245 3.73 3.840 15.41 0 0 3 4 4 14.3 8 360.0 245 3.21 3.570 15.84 0 0 3 4 5 14.7 8 440.0 230 3.23 5.345 17.42 0 0 3 4 |
如果要由大到小排列的話,則用:desc()
Use desc() to sort in descending order.
Order mpg from large to small.
| arrange(mtcars, desc(mpg)) |
|
mpg cyl disp hp drat wt qsec vs am gear carb 1 33.9 4 71.1 65 4.22 1.835 19.90 1 1 4 1 2 32.4 4 78.7 66 4.08 2.200 19.47 1 1 4 1 3 30.4 4 75.7 52 4.93 1.615 18.52 1 1 4 2 4 30.4 4 95.1 113 3.77 1.513 16.90 1 1 5 2 5 27.3 4 79.0 66 4.08 1.935 18.90 1 1 4 1 |
如果有兩個 variables 的話,會先排第一個,然後再在第一個裡面排第二個的大小順序。
| mtcars %>% arrange(cyl, gear) |
Arrange with cyl first; then within cyl, arrange gear.
上面的例子中,會先依照 cyl 的大小排列,再依 gear 的大小排列。
|
mpg cyl disp hp drat wt qsec vs am gear carb 1 21.5 4 120.1 97 3.70 2.465 20.01 1 0 3 1 2 22.8 4 108.0 93 3.85 2.320 18.61 1 1 4 1 3 24.4 4 146.7 62 3.69 3.190 20.00 1 0 4 2 4 22.8 4 140.8 95 3.92 3.150 22.90 1 0 4 2 5 32.4 4 78.7 66 4.08 2.200 19.47 1 1 4 1 |
如果想讓兩個 variables 都由大到小排列,可以分開寫,也可以合在一起。
Reorder the mtcars in descending order of mpg and displacement (disp)
|
mtcars %>% arrange(desc(carb), desc(mpg)) |
或寫成這樣:
|
mtcars %>% arrange(desc(carb, mpg)) |
|
mpg cyl disp hp drat wt qsec vs am gear carb 1 15.0 8 301.0 335 3.54 3.570 14.60 0 1 5 8 2 19.7 6 145.0 175 3.62 2.770 15.50 0 1 5 6 3 21.0 6 160.0 110 3.90 2.620 16.46 0 1 4 4 4 21.0 6 160.0 110 3.90 2.875 17.02 0 1 4 4 5 19.2 6 167.6 123 3.92 3.440 18.30 1 0 4 4 6 17.8 6 167.6 123 3.92 3.440 18.90 1 0 4 4 |
從上面可以看到是先依 carb 的大小排列,之後再依 mpg 的大小排列(最後四排當 carb 都是 4 的時候,mpg 是由大到小排列),但兩個都是由大到小。
Exercise
這邊我們用檔案資料 flights 來練習。
Q1. Which flights are the most delayed?
哪個班機起飛時間延遲最多,可以用 desc() 由大到小排列延遲的長度,第一個就是延遲最多的。下面先用 glimpse 呈現結果。
|
flights %>% arrange(desc(dep_delay)) %>% glimpse |
|
Observations: 336,776 Variables: 19 $ year <int> 2013, 2013, 2013, 2013, 2013, ... $ month <int> 1, 6, 1, 9, 7, 4, 3, 6, 7, 12, 5, 1, 2, ... $ day <int> 9, 15, 10, 20, 22, 10, 17, 27, 22, ... $ dep_time <int> 641, 1432, 1121, 1139, 845, 1100, 2321, ... $ sched_dep_time <int> 900, 1935, 1635, 1845, 1600, 1900, 810, ... $ dep_delay <dbl> 1301, 1137, 1126, 1014, 1005, 960, ... |
從上面顯示的最後一行 dep_delay 可以看到是由大到小排列。
如果不確定哪個 variable 是指延遲時間的話,可以用 ? 或 help() 的功能來查看:?flights 或是 help(flights)。
會在 R Studio 右下角的視窗中出現解釋框(如下圖,點圖可以放大),裡面寫:
|
dep_delay, arr_delay: Departure and arrival delays, in minutes. Negative times represent early departures/arrivals. |

下面是沒用 glimpse() 功能呈現,應該會比較清楚。
| arrange(flights, desc(dep_delay)) |
|
# A tibble: 336,776 x 19 year month day dep_time sched_dep_time dep_delay arr_time <int> <int> <int> <int> <int> <dbl> <int> 1 2013 1 9 641 900 1301 1242 2 2013 6 15 1432 1935 1137 1607 3 2013 1 10 1121 1635 1126 1239 4 2013 9 20 1139 1845 1014 1457 5 2013 7 22 845 1600 1005 1044 6 2013 4 10 1100 1900 960 1342 7 2013 3 17 2321 810 911 135 8 2013 6 27 959 1900 899 1236 9 2013 7 22 2257 759 898 121 10 2013 12 5 756 1700 896 1058 # ... with 336,766 more rows, and 11 more variables: # carrier &arr_delay <dbl>,lt;chr>, flight <int>, tailnum <chr>, # origin <chr>, air_time <dbl>, distance <dbl>, hour <dbl>, # minute <dbl>, time_hour <dttm> |
Q2: Which flights caught up the most time during the flight?
哪班飛機趕上的時間最多,就是說即便是延遲起飛,但是仍然準時抵達的,也就是延遲時間最短的(比預計的飛行時間短),這邊用延遲起飛的時間減掉延遲抵達的時間,再由大到小排列。
| flights %>% arrange(desc(dep_delay - arr_delay)) %>% glimpse |
|
Observations: 336,776 Variables: 19 $ year <int> 2013, 2013, 2013, 2013, 2013, 2013, ... $ month <int> 6, 2, 2, 5, 2, 7, 7, 12, 5, 11, 5, 5, ... $ day <int> 13, 26, 23, 13, 27, 14, 17, 27, 2, 13, ... $ dep_time <int> 1907, 1000, 1226, 1917, 924, 1917, ... $ sched_dep_time <int> 1512, 900, 900, 1900, 900, 1829, 1930, ... |
不用 glimpse 顯示:
| arrange(flights, desc(dep_delay - arr_delay)) |
|
# A tibble: 336,776 x 19 year month day dep_time sched_dep_time dep_delay arr_time <int> <int> <int> <int> <int> <dbl> <int> 1 2013 6 13 1907 1512 235 2134 2 2013 2 26 1000 900 60 1513 3 2013 2 23 1226 900 206 1746 4 2013 5 13 1917 1900 17 2149 5 2013 2 27 924 900 24 1448 6 2013 7 14 1917 1829 48 2109 7 2013 7 17 2004 1930 34 2224 8 2013 12 27 1719 1648 31 1956 9 2013 5 2 1947 1949 -2 2209 10 2013 11 13 2024 2015 9 2251 # ... with 336,766 more rows, and 11 more variables: # arr_delay <dbl>, carrier <chr>, flight <int>, tailnum <chr>, # origin <chr>, dest <chr>, air_time <dbl>, distance <dbl>, # hour <dbl>, minute <dbl>, time_hour <dttm> |
可以指定 catchup 為 flights,再帶入 arrange,或是用 head 只顯示前十個 carrier。
| flights %>% arrange(desc(dep_delay - arr_delay)) head(catchup$carrier, 10) |
| [1] "EV" "HA" "HA" "DL" "HA" "UA" "UA" "UA" "UA" "DL" |
如果只看資料的前十個的話,則是下面這樣。
| head(flights$carrier, 10) |
| [1] "UA" "UA" "AA" "B6" "DL" "UA" "B6" "EV" "B6" "AA" |
6. Mutate function: 設定新的 variable,新設定的 variable 會顯示在最後一欄。
Make new variables: disp_l and wt_kg
1L = 61.0237 cu.in. (cubic inch)
1kg = 2.2 lbs
檔案資料裡面的 disp 單位是 cu.in.,我們把它轉換成 L,設定其為 disp_l。同時也把裡面原本單位為磅(lbs)的 wt 換算成 kg,指定其為 wt_kg。新訂的 disp_l 和 wt_kg 會顯示在最後兩的 column。
| mtcars %>% mutate(disp_l = disp/61.0237, wt_kg = wt/2.2) |
|
mpg cyl disp hp drat wt qsec vs am gear carb disp_l wt_kg 1 21.0 6 160.0 110 3.90 2.620 16.46 0 1 4 4 2.621932 1.1909091 2 21.0 6 160.0 110 3.90 2.875 17.02 0 1 4 4 2.621932 1.3068182 3 22.8 4 108.0 93 3.85 2.320 18.61 1 1 4 1 1.769804 1.0545455 4 21.4 6 258.0 110 3.08 3.215 19.44 1 0 3 1 4.227866 1.4613636 5 18.7 8 360.0 175 3.15 3.440 17.02 0 0 3 2 5.899347 1.5636364 |
把重量換算成噸,指定其為:wt_tonnes
Include a variable for weight in tonnes (1t = 2,204.6 lbs)
| mtcars %>% mutate(wt_tones = wt / 2.2046) |
|
mpg cyl disp hp drat wt qsec vs am gear carb wt_tones 1 21.0 6 160.0 110 3.90 2.620 16.46 0 1 4 4 1.1884242 2 21.0 6 160.0 110 3.90 2.875 17.02 0 1 4 4 1.3040914 3 22.8 4 108.0 93 3.85 2.320 18.61 1 1 4 1 1.0523451 4 21.4 6 258.0 110 3.08 3.215 19.44 1 0 3 1 1.4583144 5 18.7 8 360.0 175 3.15 3.440 17.02 0 0 3 2 1.5603738 |
transmute: leave only the new variables
如果用 transmute() 的話,就只會顯示新訂的 disp_1 和 wt_kg。
| mtcars %>% transmute(disp_l = disp/61.0237, wt_kg = wt/2.2) |
|
disp_l wt_kg 1 2.621932 1.1909091 2 2.621932 1.3068182 3 1.769804 1.0545455 4 4.227866 1.4613636 5 5.899347 1.5636364 6 3.687092 1.5727273 |
Exercise
Q1. Compute speed in mph from time (in minutes) and distance (in miles)
同樣可以用 ?flights 查詢:
|
distance: Distance between airports, in miles air_time: Amount of time spent in the air, in minutes |
計算飛機的速度:mph = miles per hour
也就是飛行的距離 distance (in miles) 除以飛行的時間 air_time (in minutes)。
因為 air_time 是以分鐘為單位,所以我們必須先把它轉換成小時,也就是:air_time / 60
下面把速度指定為 speed。
| mutate(flights, speed = distance / (air_time / 60)) |
|
# A tibble: 336,776 x 20 year month day dep_time sched_dep_time dep_delay arr_time <int> <int> <int> <int> <int> <dbl> <int> 1 2013 1 1 517 515 2 830 2 2013 1 1 533 529 4 850 3 2013 1 1 542 540 2 923 4 2013 1 1 544 545 -1 1004 5 2013 1 1 554 600 -6 812 6 2013 1 1 554 558 -4 740 7 2013 1 1 555 600 -5 913 8 2013 1 1 557 600 -3 709 9 2013 1 1 557 600 -3 838 10 2013 1 1 558 600 -2 753 # ... with 336,766 more rows, and 12 more variables: # arr_delay <dbl>, carrier <chr> flight <int>, tailnum <chr>, # origin <chr>, dest <chr>, air_time <dbl>, distance <dbl>, # hour <dbl>, minute <dbl>, time_hour <dttm>, speed <dbl> |
顯示整個檔案太長,看不到後面計算出來的 speed,所以用 select() 的功能挑出我們想看的幾個 variables。如果忘記 select() 是什麼、怎麼用的話,可以看前一篇。
|
mutate(flights, speed = distance / (air_time / 60)) %>% select(carrier, distance, air_time, speed) |
| # A tibble: 336,776 x 4 carrier distance air_time speed <chr> <dbl> <dbl> <dbl> 1 UA 1400 227 370.0441 2 UA 1416 227 374.2731 3 AA 1089 160 408.3750 4 B6 1576 183 516.7213 5 DL 762 116 394.1379 6 UA 719 150 287.6000 7 B6 1065 158 404.4304 8 EV 229 53 259.2453 9 B6 944 140 404.5714 10 AA 733 138 318.6957 # ... with 336,766 more rows |
Q2. Which flight flew the fastest?
哪個班機的速度最快?可以用 arrange() 功能由大到小排列出來,跟上面一樣只挑出我們想看的幾個 variables 來看。
|
flights %>% mutate(speed = distance / (air_time / 60)) %>% arrange(desc(speed)) %>% select(carrier, distance, air_time, speed) |
|
# A tibble: 336,776 x 4 carrier distance air_time speed <chr> <dbl> <dbl> <dbl> 1 DL 762 65 703.3846 2 EV 1008 93 650.3226 3 EV 594 55 648.0000 4 EV 748 70 641.1429 5 DL 1035 105 591.4286 6 DL 1598 170 564.0000 7 B6 1598 172 557.4419 8 AA 1623 175 556.4571 9 DL 1598 173 554.2197 10 B6 1598 173 554.2197 # ... with 336,766 more rows |
Q3. 把上面班機延遲練習的 Q2,用 select() 的功能挑出我們想看的幾項出來。
|
flights %>% mutate(delay_time = dep_delay - arr_delay) %>% arrange(desc(delay_time)) %>% select(carrier, dep_time, arr_time, air_time, dep_delay, arr_delay, delay_time) |
|
# A tibble: 336,776 x 7 carrier dep_time arr_time air_time dep_delay arr_delay delay_time <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> 1 EV 1907 2134 126 235 126 109 2 HA 1000 1513 584 60 -27 87 3 HA 1226 1746 599 206 126 80 4 DL 1917 2149 313 17 -62 79 5 HA 924 1448 589 24 -52 76 6 UA 1917 2109 274 48 -26 74 7 UA 2004 2224 295 34 -40 74 8 UA 1719 1956 324 31 -42 73 9 UA 1947 2209 300 -2 -75 73 10 DL 2024 2251 311 9 -63 72 # ... with 336,766 more rows |
可以和上面排列前十的 carrier 的結果比對,看排列是不是一樣的。
7. Summarise / Summarize
功能和 mutate() 有點像,不一樣的是它會產生一個新的 data frame,並且是那欄(也就是那個 variable)所有觀察資料的總結。
| Summarise works in an analogous way to mutate, except instead of adding columns to an existing data frame, it creates a new data frame. This is particularly useful in conjunction with ddply as it makes it easy to perform group-wise summaries. |
例如算出 mpg 的平均值,可以用 mean() 的功能這樣算:
| mean(mtcars$mpg) |
| [1] 20.09062 |
如果用 mutate() 的話會是這樣:
| mutate(mtcars, mean(mpg)) |
|
mpg cyl disp hp drat wt qsec vs am gear carb mean(mpg) 1 21.0 6 160.0 110 3.90 2.620 16.46 0 1 4 4 20.09062 2 21.0 6 160.0 110 3.90 2.875 17.02 0 1 4 4 20.09062 3 22.8 4 108.0 93 3.85 2.320 18.61 1 1 4 1 20.09062 4 21.4 6 258.0 110 3.08 3.215 19.44 1 0 3 1 20.09062 5 18.7 8 360.0 175 3.15 3.440 17.02 0 0 3 2 20.09062 |
同樣是算出 mean,但它會放在最後一欄,所以所有數值都一樣,因為它是全部觀察算出來的平均值。
如果用 summarise() 的話則是這樣,以一個 data frame 的樣式呈現出來:
| summarise(mtcars, mean(mpg)) |
|
mean(mpg) 1 20.09062 |
也可以幫平均值指定一個名稱 mpg_mean,直接寫在裡面即可。
| summarise(mtcars, mpg_mean = mean(mpg) |
| mpg_mean 1 20.09062 |
也可以設定三個,例如把 mpg 的平均值設為 mean_mpg,把重量的中間值設為 median_wt,然後把這兩者的比例設為 ratio。
找出中間值用的功能是:median()
| mtcars %>% summarise(mean_mpg = mean(mpg), median_wt = median(wt), ratio = mean_mpg/median_wt) |
|
mean_mpg median_wt ratio 1 20.09062 3.325 6.042293 |
也可以全部寫在一行,下面同時把 mpg 的平均值直接設為 mpg,wt 的中間值設為 wt,把兩者的比例設為 ratio。
| summarise(mtcars, mpg = mean(mpg), wt = median(wt), ratio = mpg/wt) |
|
mpg wt ratio 1 20.09062 3.325 6.042293 |
除了算全部的平均值外,也可以分組算,例如算出 cyl 裡各種觀察值的平均值。下面先用 levels() 看 cyl 裡面有哪幾種,因為 cyl 的觀察值是數字,需要先把它換成 factor。
| levels(factor(mtcars$cyl)) |
| [1] "4" "6" "8" |
由上面結果可以看出 cyl 的觀察值有三種:4, 6, 8。如果要看各個的 mpg 和重量平均值,可以用 group_by() 的功能。
|
mtcars %>% group_by(cyl) %>% summarise(mean_mpg = mean(mpg), median_wt = median(wt)) |
|
# A tibble: 3 x 3 cyl mean_mpg median_wt <dbl> <dbl> <dbl> 1 4 26.66364 2.200 2 6 19.74286 3.215 3 8 15.10000 3.755 |
也可以在一組裡面再分組算平均值,例如先用 am 分組後,在再裡面用 cyl 分組,先分的那個放前面,所以語法會是:group_by(am, cyl)
|
mtcars %>% group_by(am, cyl) %>% summarise(mpg = mean(mpg), wt = median(wt)) |
| # A tibble: 6 x 4 # Groups: am [?] am cyl mpg wt <dbl> <dbl> <dbl> <dbl> 1 0 4 22.90000 3.1500 2 0 6 19.12500 3.4400 3 0 8 15.05000 3.8100 4 1 4 28.07500 2.0375 5 1 6 20.56667 2.7700 6 1 8 15.40000 3.3700 |
因為用 summarise() 算出來的結果本身就會轉換成一個 data frame,所以可以把這個 data frame 指定一個名稱,例如為 cars_am_cyl,這樣之後想要看的時候便不需要打全部的語法,只要叫出 cars_am_cyl 即可看。
| cars_am_cyl <- mtcars %>% group_by(am, cyl) %>% summarise(mpg = mean(mpg), wt = median(wt)) cars_am_cyl |
因為 cars_am_cyl 已經是一個 data frame,也可以直接用它來做運算,因為上面設定時已經先用 am 分好組了,所以算平均值的時候會依這個分。
| cars_am_cyl %>% summarise(mpg = mean(mpg), wt = median(wt)) |
| # A tibble: 2 x
am mpg wt <dbl> <dbl> <dbl> 1 0 19.02500 3.44 2 1 21.34722 2.77 |
也可以相反設試試看,先用 cyl 分組後再依 am 分組運算,也就是 group_by(cyl, am),然後和上面比較有何不同。
| cars_cyl_am <- mtcars %>% group_by(cyl, am) %>% summarise(mpg = mean(mpg), wt = median(wt)) cars_cyl_am |
| # A tibble: 6 x 4 # Groups: cyl [?] cyl am mpg wt <dbl> <dbl> <dbl> <dbl> 1 4 0 22.90000 3.1500 2 4 1 28.07500 2.0375 3 6 0 19.12500 3.4400 4 6 1 20.56667 2.7700 5 8 0 15.05000 3.8100 6 8 1 15.40000 3.3700 |
因為上面是先用 cyl 分組,所以接下來用 cars_cyl_am 來做運算的話,會算出 cyl 裡各組的平均值。
| cars_cyl_am %>% summarise(mpg = mean(mpg), wt = median(wt)) |
| # A tibble: 3 x 3 cyl mpg wt <dbl> <dbl> <dbl> 1 4 25.48750 2.59375 2 6 19.84583 3.10500 3 8 15.22500 3.59000 |
Exercise
Q1. Compute the minimum and maximum displacement for each engine type (vs) by transmission type (am)
在各個引擎的種類中依 am 分組後,找出其中的最大值和最小值,因此要先依引擎分類,再在其中依 am 分組,也就是:group_by(vs, am)
最大值和最小值的功能為:max() 和 min()
| mtcars %>% group_by(vs, am) %>% summarise(min = min(disp), max = max(disp)) |
| # A tibble: 4 x 4 # Groups: vs [?] vs am min max <dbl> <dbl> <dbl> <dbl> 1 0 0 275.8 472 2 0 1 120.3 351 3 1 0 120.1 258 4 1 1 71.1 121 |
Q2. Which destinations have the highest average delays?
| flights %>% group_by(dest) %>% summarise(avg_delay = mean(arr_delay, na.rm = TRUE)) %>% arrange(desc(avg_delay)) |
| # A tibble: 105 x 2 dest avg_delay <chr> <dbl> 1 CAE 41.76415 2 TUL 33.65986 3 OKC 30.61905 4 JAC 28.09524 5 TYS 24.06920 6 MSN 20.19604 7 RIC 20.11125 8 CAK 19.69834 9 DSM 19.00574 10 GRR 18.18956 # ... with 95 more rows |
8. Boxplot
接下來試著畫箱圖,如果想畫出在 cyl < 8 的資料中,mpg 對 cyl 的反應,也就是:
|
x-axis: cyl < 8 y-axis: mpg, response to cyl |
boxplot 的語法是:boxplot(formula, data = )
formula 是指兩軸(也就是兩個 variables)的關係,在這裡是: y ~ x
表示 Y-axis 對 X-axis 的反應,所以也就是:boxplot(y ~ x, data = )
最後用 subset = 的功能挑出你要的,在這個例子裡就是 cyl < 8。
| boxplot(mpg ~ cyl, data = mtcars, subset= cyl < 8) |

也可以用 %>% 分開寫。
You can use . as a placeholder when the “data” argument is in the second position.
| mtcars %>% filter(cyl < 8) %>% boxplot(mpg ~ cyl, data = . ) |
在上面的語法裡,當前面已經表示過用的資料是 mtcars 的時候,後面在 boxplot() 裡面 data 的部分就可以用 "." 替代,也就是:data = .
也可以用 ggplot2 來畫圖,用 ggplot 裡的 geom_boxplot() 畫的話,語法就是這樣:
| ggplot(mtcars %>% filter(cyl < 8), aes(factor(cyl), mpg)) + geom_boxplot() |
因為 cyl 是數字,所以要需要先把它變成 factor,想更了解的話可以參考這篇:R | ggplot: Point plot & Box plot
也可以先把畫圖要用的資料用 subset() 的功能挑出來,也就是 cyl < 8 的部分。我們可以把挑出來的部分指定成一個新的 data frame,下面我們把指定其為 cyl_sub,然後再用它來畫圖。
|
cyl_sub <- subset(mtcars, cyl < 8) ggplot(cyl_sub, aes(factor(cyl), mpg)) + geom_boxplot() |

好了,這篇就先到這吧。
訂閱:
文章 (Atom)